MolecularDynamics
物理実験1(分子動力学法)
3年生前期の物理実験1の実験テーマのひとつです。
物理学コースの学生は必ず「分子動力学法」をおこなう必要があります。
計算科学コースの学生は受けることができません(計算実験で分子動力学に関するさらに詳しい実験があります)
- 2018年度は山口直也さんがティーチングアシスタントです。
- 2018年度以降は小幡先生が担当されます。
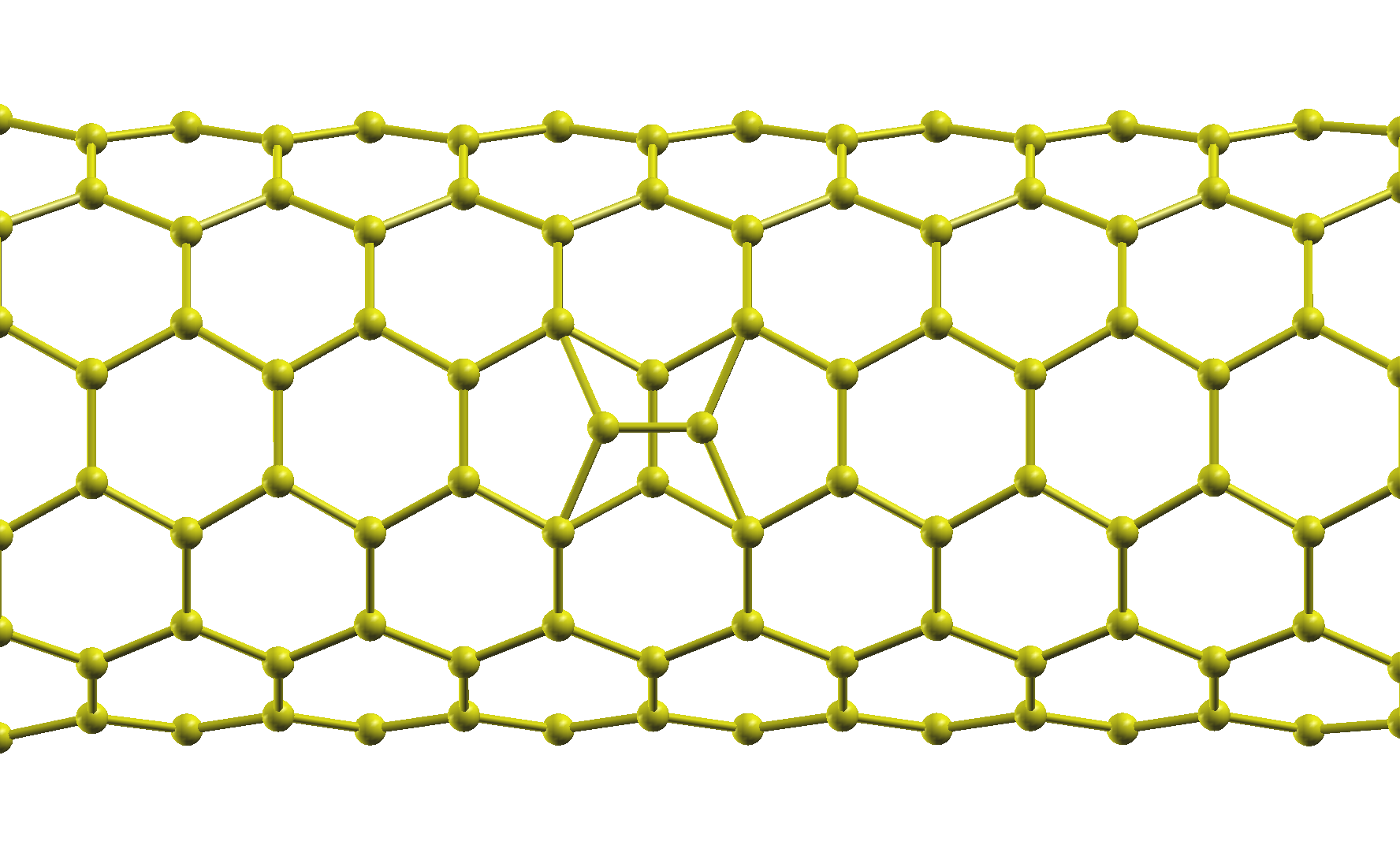
炭素からなる物質の例
カーボンナノチューブ(6,6)のStone-Wales 欠陥
最終更新時間:2019年07月11日 13時48分52秒
レポートに関する注意事項
- 提出はメール(pdfファイルが望ましい)とプリントしたもの両方を提出願います
(提出締切に印刷が間に合わない場合はpdfだけでもOKです。ただし、手渡しの代わりに居室に口頭で
メールを送りました、と伝えにくること)
- 合格になるまで1週以内程度に再提出を続けてください。
- 再提出の無い場合は最低点になる可能性があります。
- チェックリストchecklist.pdf(608)/checklist.doc(426)をチェックして提出してください。
- latex:report_temp.tex(899)(report_temp_win.tex(402)]:windows用)か, word :report_temp2010.doc(362)のテンプレートを使用してください。
- コンパイル例:report_temp.pdf(469)
- 一回目は未完成のものでも良いので提出してください。
実験に関する注意事項
- 不明な点は積極的に質問すること.
- 質問すること自体, 実験を迅速に進めていく上で重要なポイントで、評価対象にもなり得ます。
内容
配布資料
配布資料に書いてある, 1回目, 2回目等は目安です。実験のペースは各自で決めてください.
print20130424.pdf(647)
fortran77の練習
- Fortran入門(超入門)
- 練習プリントexercise.pdf(614)
report_temp.pdf(469)
fortranの注意事項
- 行番号は左から5列目に一桁がくるように書く。 100なら3,4,5列に書く。
- 長い行の場合&を打つ、&は6列目。
tbmdコンパイル時にファイルを変更する点
- rannum.fの16行目をコメントアウト
- Makefileの6行目のg77をgfortranへ変更
初期構造の作り方
絵を書いたり、プログラムを作成したり工夫が求められる。
座標生成プログラムの例
(ccluster.datを出力する。これらの例を参考に, グラフェンの破片など、自分のプログラムを作成してみよう)
- 直鎖
data_chain.f(546)
- 環状
data_circle.f(485)
シェルスクリプトを作成する等で、自動的に座標を生成し、最後のエネルギーをとってきて、凝集エネルギーを
原子数について1-60までを一度に計算することも可能である。
座標をもらってくる
- カーボンナノチューブ生成ウェブページ
http://turin.nss.udel.edu/research/tubegenonline.html
- C60 isomer
http://www.nanotube.msu.edu/fullerene/fullerene-isomers.html
C20-C720までの座標がまとめてある。xyzファイルをダウンロードできる(angstromeなのでxyzのみ抽出して0.1倍する必要がある
XCrysDen以外を使う
- Vesta
windows, mac, linux版がある。
xyzファイルを読み込める。
http://jp-minerals.org/vesta/jp/
tbmd全般
mdinput.dat
- 原子数を増やしたりする場合や、安定構造を計算する上で
- 変更するファイルその1:mdinput.dat
5行目の左側の数字:原子数 10行目の摩擦の係数を0.01ぐらいに(配布ファイルは0.008だから、そのままでも良い)
- 変更するファイルその2:ccluster.dat
ccluster.dat 1行目に原子数 ccluster.datの下に原子数の数だけ座標 x,y,z (単位はnm)
tbmd.fの変数など
iatomは総原子数 N_atomまで
- coord0(iatom,1): iatom番目の原子のx座標
- coord0(iatom,2): iatom番目の原子のy座標
- coord0(iatom,3): iatom番目の原子のz座標
- forceは力, veloは速度で同様。
力の出力と表示
- 配布プリント 「7.5 原子位置の出力」で
write(16,100)coord0(i_atom,1)*fac,coord0(i_atom,2)*fac,
& coord0(i_atom,3)*fac
100 format('C ', 3(1X,E15.6))
の部分を
write(16,100)coord0(i_atom,1)*fac,coord0(i_atom,2)*fac,
& coord0(i_atom,3)*fac,
& force(i_atom,1)*fac, force(i_atom,2)*fac, force(i_atom,3)*fac
100 format('C ', 6(1X,E15.6))
として, XCrysDenで「Display」--->「force」とすると矢印が表示される。
矢印が大きすぎる場合, プログラムのfacをfac2=1/1000等変更して定義して, forceにかけて出力する、
もしくは XCrysDenで「Modify」--->「Force Settings」のLength Factorを小さくする等すればよい。
原子数が60以上の計算をする場合
プログラム中の配列宣言を変更する必要があります。
自分でどこを変更すれば良いか、探してみよう。
出力について
出力されるエネルギーは原子あたりのエネルギーである。
書式
- f15.6
- e.15.6
など自分で調べること
XCrysDen
fort.16等のxyz形式のファイルを読み込んで表示する場合,
xcrysden --xyz fort.16
とするとよい.
フーリエ変換のヒント
例えばプリントの式通りに書いてみる.
omeg=dble(iomega)*1.0d0/100.0d0
A(1,1,iomega)=A(1,1,iomega)+coord0(1,1)*dt
& *cos(tpi*dble(i_time_step)*omeg*dt)
これをiomegaで100までループで回すとかして, 時間のループの中にいれる.
(配列の初期化や宣言が別途必要である)
全ステップ終了後にomega毎にデータをwriteする.
ただし、原点をどう置くかに注意を要する
(原子の変位がcos波になるように座標の原点を移動する)。
テキストにあるように座標ではなく速度を用いてフーリエ変換するのがよい。
gnuplotからjpegファイルに落として、latexへ
通常はepsかpsファイルを用いる(その場合はbbファイルは要らない
例:sin(x)をjpgファイルに
gnuplotで
set term jpeg set output 'sin.jpg' plot sin(x)
latexにjpgファイルを貼るにはbbファイルが必要である.
ebb sin.jpg
とするとsin.bbというファイルができるので, sin.bbがsin.jpgと
同じディレクトリにあれば, psやepsのファイルと同様にlatexに貼ることができる.
数値の単位
レポートのテンプレート
latex
下記の3つのファイルをダウンロードして, report_temp.texをコンパイルする.
H5O2plus.pdf(397)
report_temp.tex(899)
H5O2plus.bb(253)
word
- report_temp2010.doc(362)
レポート提出前チェックリスト
- checklist.doc(426)
cnt_sw.png(279)
参考文献など
checklist.pdf(608)
report_temp_win.tex(402)
print.pdf(1070)
print20130424.pdf(647)
tbmd.f(290)